



我院张利达副教授利用生物大数据解析水稻蛋白质互作组
近日,植物学领域国际著名期刊《The Plant Journal》在线发表了题为“A computational interactome for prioritizing genes associated with complex agronomic traits in rice”的研究论文。该研究报道了水稻蛋白质互作组最新研究成果,我院张利达副教授为本文的通讯作者,硕士生刘诗薇与刘奕慧为并列第一作者。这是继不久前张利达副教授在植物学领域另一国际著名期刊《Plant physiology》上发表拟南芥全基因组蛋白质互作网络(Genome-Wide Inference of Protein-Protein Interaction Networks Identifies Crosstalk in Abscisic Acid Signaling)后又一次基于生物大数据挖掘所取得的研究成果。
蛋白质-蛋白质相互作用对于几乎所有的细胞过程都至关重要,蛋白质相互作用研究能够从分子水平上揭示蛋白质的功能,帮助揭示生长发育、新陈代谢、分化和凋亡等细胞活动规律。目前虽然已有一些蛋白质相互作用的高通量实验方法,但由于实验手段耗费大量的人力和物力,蛋白质相互作用的大规模解析还远未在植物学研究中普及。本研究提出了一种基于机器学习的水稻蛋白质互作组预测新方法,该方法结合了蛋白质三维结构与功能信息,极大地提高了蛋白质相互作用预测的准确性与覆盖率,各项可比指标均大大优于现有预测方法。利用新方法所建立的水稻蛋白质互作组可为水稻分子生物学研究提供重要的数据资源,尤其对水稻复杂农艺性状的分子解析具有重要的理论和实践指导意义。
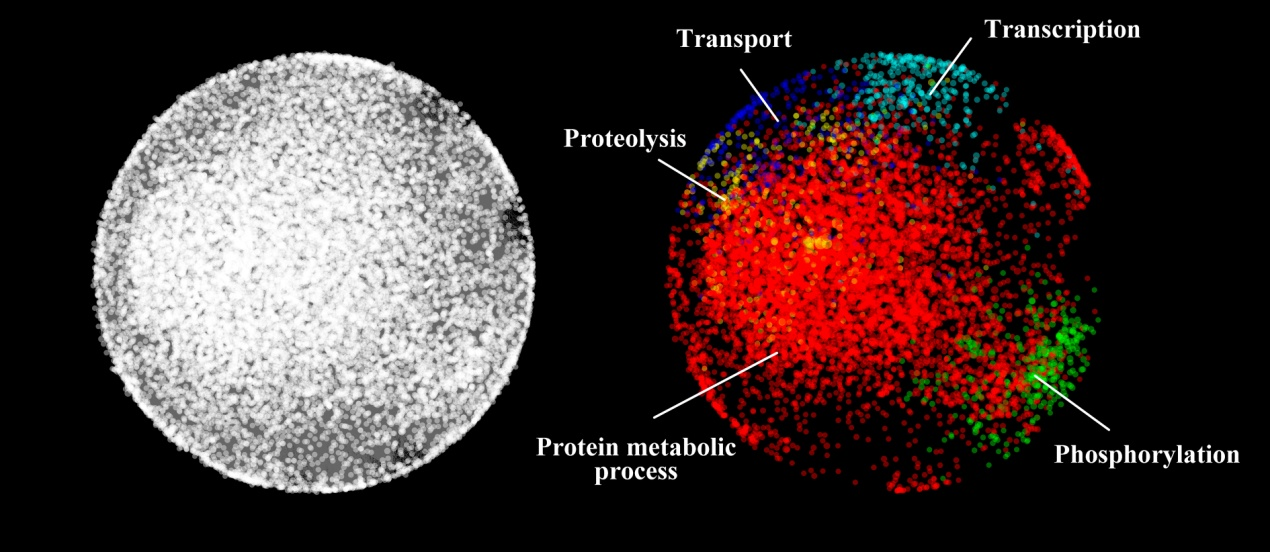
水稻蛋白质互作组概览图
该研究得到了国家自然科学基金项目(31371229)与国家973项目(2015CB755702)的资助。该研究的顺利实施得到了新葡的京集团35222vip高性能计算中心的支持。
文章链接:http://onlinelibrary.wiley.com/doi/10.1111/tpj.13475/full
http://www.plantphysiol.org/content/171/2/1511.full
Copyright 2016 新葡的京集团35222vip(股份)有限公司 -Official Entrance 访问旧版
地址:上海市闵行区东川路800号 电话:021 - 34205866 备案:沪交ICP备20101025

 loading......
loading......